借助一系列简介、采访、悼文以及相关原始文献,文章系统地回顾了查尔斯·戴维·阿利斯 (C. David Allis) 传奇的一生,梳理了表观遗传学开拓者、染色质生物学先驱查尔斯·戴维·阿利斯影响深远的贡献。其中包含4项原创研究贡献:发现第一个组蛋白乙酰基转移酶;揭示组蛋白H3第10位丝氨酸磷酸化 (H3S10ph) 与细胞分裂的关系;参与发现第一个组蛋白位点特异性的甲基转移酶;推动组蛋白变体和“致癌组蛋白 (oncohistone)”领域的进展。一项理论贡献:提出“组蛋白密码 (histone code)”的假说。两项对表观遗传学界的贡献:作为发起人之一,参与对组蛋白修饰酶的系统命名;作为主编之一,编撰Epigenetics专著。
撰文 | 徐鹏(耶鲁大学医学院)
查尔斯·戴维·阿利斯 (C. David Allis, 1951. 3. 22-2023. 1. 8) 是公认的表观遗传学和染色质生物学的先驱。他勤耕不辍且富有传奇色彩的一生极大地推动了表观遗传学和染色质生物学的进展,并培养了一大批优秀的科学家。他的猝然离世是生物医学界的重大损失,人们纷纷悼念这位备受尊重的杰出科学家。2023年2月3日至2月16日期间,Nature、Science、Cell杂志相继发文悼念[1-3],2月27日C. David Allis实验室前成员在Nature Genetics发表悼文[4]。然而,寥寥几篇悼文远不足以概括C. David Allis传奇的一生。借2005年他当选美国科学院院士后PNAS的介绍 (2006年发表) [5]、2015年的一篇采访[6]、前文提到的4篇悼文[1-4]以及一系列相关原始文献,本文尽可能详尽地回顾C. David Allis不断书写传奇的一生,并系统梳理其影响深远的原创研究贡献、理论贡献以及对表观遗传学界的贡献。C. David Allis独特的科研品味、宽广的胸襟以及乐于助人的品格使他的形象愈发高大。
1
追随兴趣,从辛辛那提到纽约
C. David Allis全名Charles David Allis,他的同事和好友们称呼其昵称Dave,本文使用Allis。
Allis于1951年出生于美国俄亥俄州的辛辛那提,1969年本科就读于家乡的辛辛那提大学生物学专业。大四前夕,他正在找做毕业论文的实验室,准备毕业后去医学院继续深造。Steven Keller教授建议他在下定决心去医学院之前,去“一个真正的科学实验室 (a real lab)”。Keller给Allis牵线, 将其介绍给Michael Bharier教授。而正是在大四那年,在Bharier实验室,他对基础生物学产生了兴趣。后来回想起来时,Allis对Keller和Bharier心怀感激: “他们看到了我内心的科学火种,并点燃了它。”
1973年,他以“最优等 (summa cum laude) ”的成绩本科毕业。不顾朋友和父母的期许,他彻底忘掉此前的医生梦,进入印第安纳大学伯明顿分校Anthony Mahowald实验室攻读博士学位,尽管Mahowald于一年前才搬到印第安纳大学建立自己的实验室。在Mahowald实验室,Allis主要研究果蝇发育。早期果蝇胚胎形成两种类型的生殖颗粒:细胞质中促进原始生殖细胞形成的“极性颗粒”和细胞核中促进原始生殖细胞有丝分裂的核颗粒。Allis的研究聚焦于果蝇的极细胞(生殖细胞)和极性颗粒。值得一提的是,Mahowald后来于1994年当选美国科学院院士,而且当时他已是芝加哥大学分子遗传和细胞生物学系的系主任。
1978年Allis取得生物学博士学位后,进入纽约罗彻斯特大学,在Martin Gorovsky实验室进行博士后训练。在那里他开始专注于研究染色质生物学(chromatin biology)。在与Gorovsky教授讨论后,Allis被他的个性和用四膜虫这种独特的模式生物研究染色质的优势所吸引。在那个年代,染色质生物学是绝对的冷门。尽管一些科学家们利用四膜虫做出多项获得诺贝尔奖的工作,但在当时四膜虫并不被广泛看好。
1981年Allis进入位于休斯顿的贝勒医学院担任助理教授,继续深入研究四膜虫,深耕染色质生物学。尽管在当时,他的研究领域 (染色质生物学) 和所使用的模式生物 (四膜虫) 都比较冷门,但他得到了主任Salih Wakil的大力支持。Allis非常自信:“我从来不觉得自己是个二流的科学家 (I never felt like a second-class scientist)。” 他在贝勒医学院工作了9年,在此期间逐步晋升为生物化学和细胞生物学的副教授和正教授。而从1978年博士后开始到1996年发现第一个组蛋白乙酰化酶,他养了18年四膜虫,也深耕染色质生物学18年。
在贝勒医学院工作了9年后, Allis搬到纽约州的雪城大学,并于1995年回到了他做博士后的罗彻斯特大学,而正是在这里,他取得突破性进展——发现了第一个组蛋白乙酰基转移酶 (histone acetyltransferase, HAT) 。3年后,他搬到弗吉尼亚大学科学健康中心,在这里他和博士后Brian D. Strahl提出了足以载入科学史册的“组蛋白密码 (histone code) ”假说。
毫无疑问,1996年发现第一个组蛋白乙酰基转移酶的工作奠定了Allis的历史地位,而2000年的“组蛋白密码”假说,实现了从原创研究到理论的跃迁,使Allis的贡献和影响更加广泛和深远。2003年,他又一次搬家,此次的目的地是洛克菲勒大学,并在这里度过了自己余生的科研时光 (Allis的人生轨迹参见图1)。在洛克菲勒大学的这20年中,他依然保持高水平的研究产出,Allis在某种意义上已经变成了表观遗传领域的带头人。Allis的工作广受认可,获得了一系列重量级荣誉,包括2005年当选美国科学院院士,2007年获加拿大盖尔德纳奖,2014年获日本奖,2015年获科学突破奖,2018年与MichaelGrunstein共享拉斯克奖。拉斯克奖的多位获得者后来也荣获了诺贝尔奖,因此被称为“诺贝尔奖的风向标”。实际上,近些年表观遗传研究获得诺贝尔奖的呼声越来越高。毫无疑问,如果诺贝尔奖颁发给表观遗传领域的学者,Allis将是最热门的候选人。
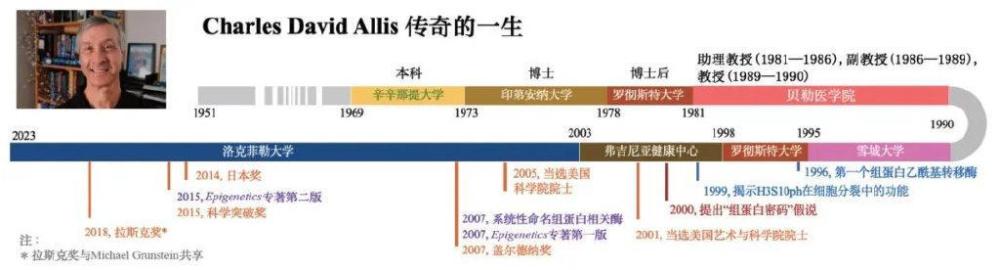
图1 系统回顾Charles David Allis的一生 (颜色条长度可反映时间长度)
2
原创研究贡献
2.1 发现第一个组蛋白乙酰基转移酶
DNA 并不是独立存在的,DNA缠绕在组蛋白八聚体上形成染色质的基本单元——核小体。20世纪60年代,Vincent Allfrey等在组蛋白修饰方面的研究提示组蛋白乙酰化与基因活性紧密相关[7]。既然早期的一些研究已经提示了组蛋白乙酰化与基因活性相关,那么一个关键问题是,组蛋白乙酰化是如何形成的?
20 世纪90 年代,很多实验室开始竞相寻找可以修饰组蛋白的酶。要鉴定修饰组蛋白乙酰化的酶并不容易,需克服技术上的困难。经过多年的累积,1995年Allis和他的研究生James Brownell发现组蛋白乙酰化的活性可以在十二烷基硫酸钠 (sodium dodecyl sulfate, SDS) 处理后依然存在,由此开发了一种活性凝胶实验 (an activity gel assay) 来鉴定组蛋白乙酰化酶,并发现了具有组蛋白乙酰基转移酶活性的催化亚单元p55[8]。一年后的1996年,Allis等克隆了p55基因的序列,发现四膜虫的p55是芽殖酵母中转录共激活因子Gcn5的同源类似物[9],提示组蛋白乙酰化与基因活性存在联系。至此,寻找组蛋白乙酰基转移酶的竞赛以Allis获胜而告终。此后,其他组蛋白乙酰化酶相继被鉴定,包括TAF1[10]、CBP/p300[11-12]、SRC-1[13]、PCAF[14]等。
Allis之所以成功,很重要的原因之一是使用了四膜虫这种模式生物。为什么是四膜虫?四膜虫有两个独立的核 (图2):大核 (macronucleus, MAC) 中有体细胞基因组,具有转录活性,染色质呈现高乙酰化水平;小核 (micronucleus, MIC) 中储存生殖基因组,转录沉默,缺少染色质修饰。大核中有较高水平的乙酰化,这提示大核中可能有较高水平的组蛋白乙酰基转移酶。如果用其他细胞或模式生物,可能得不到很好的结果。
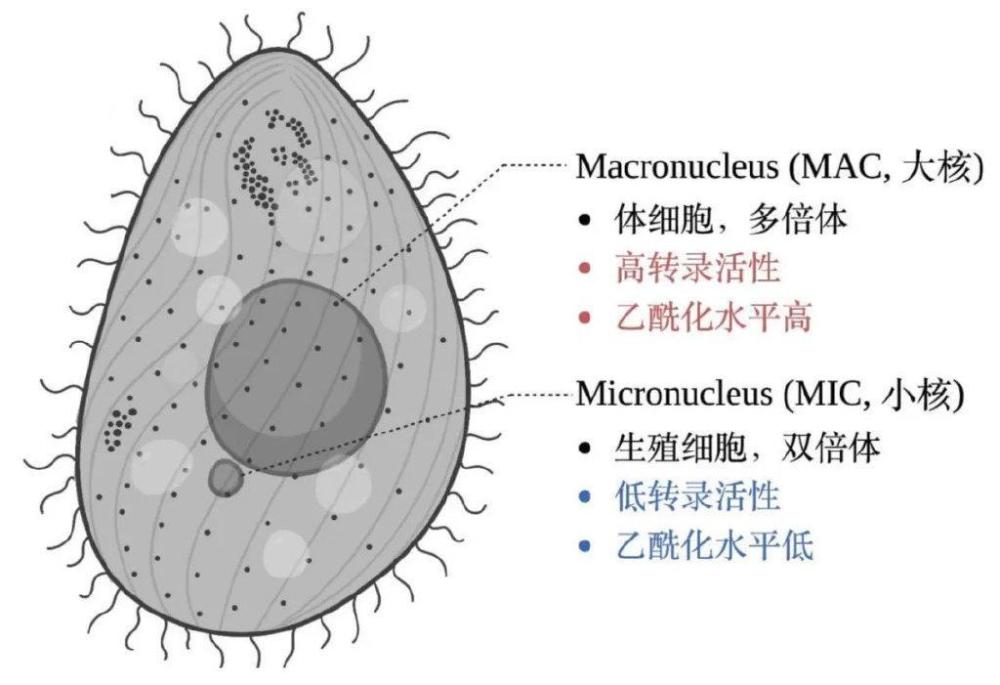
图2 四膜虫的大核和小核 (使用BioRender绘制)
在关于HAT p55的文章发表一个月后,化学生物学家Stuart Schreiber就鉴定了第一个组蛋白去乙酰化酶 (histone deacetylase, HDAC)。巧合的是,这个HDAC是酿酒酵母中的转录抑制因子Rpd3的同源类似物[15],这次是将组蛋白去乙酰化与基因转录抑制联系起来!这两篇间隔仅一个月的突破性文章,深刻地改变了我们对于DNA表达调控的理解。随之而来的模型是:基因激活涉及组蛋白乙酰基转移酶HAT,而基因抑制涉及组蛋白去乙酰化酶HDAC——HAT和HDAC就好似基因调控的开关。现代染色质生物学由此发端。
2.2 揭示细胞分裂过程中组蛋白H3第10位丝氨酸磷酸化的功能
组蛋白磷酸化是Allis实验室长期耕耘的方向。1993年Allis研究团队发现,cAMP蛋白激酶可磷酸化四膜虫处于有丝分裂状态的小核中的接头组蛋白 (linker histone) [16]。1998年,他们进一步发现,四膜虫的有丝分裂和减数分裂过程中,组蛋白H3第10位丝氨酸的磷酸化 (H3S10ph) 与染色质的压缩紧密相关[17]。1999年,Allis研究团队的突破性发现证实了H3S10ph对细胞分裂过程中染色质的压缩和分离是必需的[18]。此外,在研究中Allis团队还制备了H3S10ph的抗体,而且非常慷慨地分享抗体给其他研究者。时至今日,组蛋白H3S10ph已经成为有丝分裂过程中最重要的标志之一。
2.3 参与发现第一个组蛋白甲基化酶
在发现第一个组蛋白乙酰化酶以后,Allis鼓励实验室成员用DNA 甲基化的辅因子S-腺苷甲硫氨酸 (S-adenosyl methionine, SAM) 替换乙酰化反应的辅因子乙酰辅酶A,重复体外的HAT试验 (histone acetyltransferases assay),来探究四膜虫大核中是否也存在DNA甲基化的过程。
在1999年的科罗拉多的美国实验生物学会联合会 (Federation of American Societies for Experimental Biology, FASEB)会议上,Allis与Thomas Jenuwein邂逅并由此开启合作。一年后Jenuwein等发现了首个组蛋白甲基转移酶——SUV39H1,它可催化组蛋白H3K9的甲基化,其催化结构域位于保守的SET结构域中[19],而Allis也参与了此项研究。Allis还开玩笑说:“其实名字就提示SUV39H1可催化H3K9的甲基化,3提示组蛋白H3,9提示赖氨酸K9。” [5]
此前在果蝇中的研究已经将Su(var)3–9与位置效应花斑 (position-effect variegation) 和异染色质联系起来。此外,一些包含SET结构域的蛋白也与果蝇发育过程中的Hox基因表达状态相关,包含SET结构域的Trithorax (TrxG)参与激活状态,而Polycomb (PcG) 参与沉默状态[20]。因此,包含SET结构域的组蛋白甲基化酶SUV39H1的发现意义重大,这也激励更多的研究者投身到组蛋白甲基化相关酶的鉴定及其生物学功能研究中。 一个重大突破是2004年哈佛大学施扬团队发现了首个组蛋白去甲基化酶LSD1[21],这说明组蛋白甲基化与其他众多组蛋白修饰一样,也是可逆和动态的,由此开启了组蛋白甲基化动态调控研究的新篇章。
2.4 推动组蛋白变体和“致癌组蛋白”领域的进展
1996-2004年期间,组蛋白乙酰基转移酶、去乙酰化酶、甲基转移酶和去甲基化酶的相继发现,掀起了组蛋白修饰相关酶鉴定和生物学功能研究的浪潮。实际上,组蛋白本身存在多种变体 (histone variant),而且组蛋白及其变体上存在多种突变 (histone mutation)。这些突变有什么生物学功能呢?2012年的几项研究首次在儿童/青少年多形性胶质母细胞瘤 (glioblastoma multiforme, GBM) 和儿童扩散型内因性脑桥神经胶质瘤 (pediatric diffuse intrinsic pontine glioma, DIPG) 中,发现了高频出现的组蛋白H3.3等变体以及相关伴侣蛋白编码基因的突变[22-24],这提示组蛋白突变可能与肿瘤的发生密切相关。
在2012年突破性研究成果出来前,Allis团队也一直在研究组蛋白变体,而且已经做出了一系列重要的成果,包括揭示了组蛋白H3. 3全基因组的分布图谱,发现ATRX和DAXX伴侣蛋白对H3.3的调控[25-26]。因此,当2012年组蛋白变体突变的研究出来后,Allis立即意识到这几项研究的重要性。通过与临床医生和癌症生物学家合作,Allis团队率先阐明了这些组蛋白突变的机制[27]。组蛋白及其变体的突变与肿瘤发生密切相关,因此被称为 “致癌组蛋白 (oncohistone)”。2019年Allis团队系统性地绘制了肿瘤中“致癌组蛋白”的图谱[28]。现如今,研究者们正致力于探究组蛋白及其变体、组蛋白修饰相关酶的突变与肿瘤等疾病之间的关系。
Allis实验室前成员们在追忆文章中提到,最早是Allis将组蛋白及其变体的突变创造性地称为 “致癌组蛋白”[5]。虽然未有明确文献证实这一贡献,但毫无疑问的是,Allis团队关于组蛋白变体以及“致癌组蛋白”的一系列研究进一步拓展了人们对组蛋白调控的认知。
3
理论贡献:提出“组蛋白密码”假说
在2000年时,科学家们已经发现组蛋白尾巴上存在多种化学修饰,如乙酰化、磷酸化、甲基化和ADP-核糖基化等。随着更多种组蛋白修饰及其修饰酶的发现,Allis开始思考如何解释这些组蛋白修饰及其生物学功能。
2000年,彼时还在弗吉尼亚大学健康科学中心的Allis和实验室博士后Brian D. Strahl在Nature发文,提出了“组蛋白密码”的假说。他们认为:在一个或多个组蛋白尾巴上的不同组蛋白修饰,能够相继地或组合地发挥作用,形成“组蛋白密码”;这套密码可被其他蛋白读取,并导致不同的下游事件[29]。一年后,Allis与合Jenuwein在Science发表综述,更为系统地阐述了“组蛋白密码 ”的[30]。如今,“组蛋白密码”成为表观遗传学领域最常被引用的理论之一,大量的研究仍在这一框架下进行。
这一概念具有野心和远见:DNA双螺旋结构的解析让我们认识到遗传密码储存在DNA中;而基于组蛋白及其修饰的调控,如果是另一套密码,在某种程度上它将是可以与基于DNA双螺旋结构的遗传密码相媲美的成就。尽管“组蛋白密码”的一些方面还存在争议[31],但Allis认为这个概念的提出并不意味着削弱其他基因调控机制的重要性亦或取而代之,而是提供了一种较为通俗的表述,阐述组蛋白翻译后修饰如何拓展其调控功能。
4
对表观遗传学界的贡献
4.1 倡议系统命名组蛋白修饰酶
2007年, Allis联合表观遗传学领域的一批杰出学者,对染色质修饰酶进行系统性命名[32],比如将组蛋白赖氨酸乙酰基转移酶统一命名为KAT,将组蛋白赖氨酸甲基转移酶统一命名为KMT,将组蛋白去甲基化酶统一命名为KDM。这一系统命名得到超过80位顶尖科学家的支持,极大地方便了后续的研究和交流。
Allis实验室的前成员们在追忆Allis的文章中还提到,Allis将添加和去除组蛋白修饰的酶分别称为“书写子 (writer) ”和“擦除子 (eraser) ”,而将能识别特定组蛋白修饰的蛋白称为“识别子 (reader) ”[5]。这种通俗易懂的类比 (图3) 生动形象地解释了组蛋白修饰发生、去除和识别的过程,为理解其生物学功能以及诠释“组蛋白密码”提供了很好的语言,如今这套类比已被广泛接受。尽管Allis在2004年的一篇文章中已明确提到“书写和阅读‘组蛋白密码’ (writing and reading the histone code) ”[33],但至于是否完全归功于Allis,未有充分的文献证实。
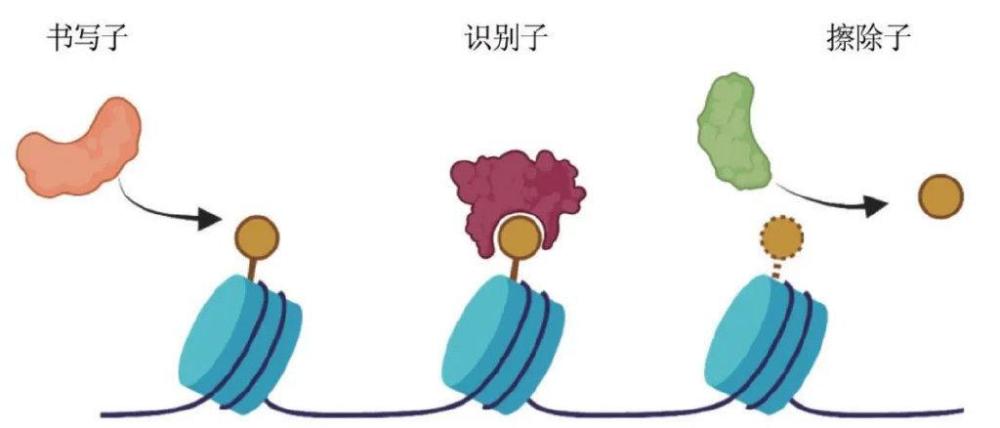
图3 组蛋白修饰的书写子(writer)、擦除子(eraser)和识别子(reader)(使用BioRender绘制)
4.2 Epigenetics专著
自1996年发现第一个组蛋白乙酰基转移酶和去乙酰化酶以来,表观遗传获得越来越多的关注,表观遗传研究呈井喷之势 (图4)。在这一背景下,2007年Allis联合Jenuwein和Danny Reinberg,共同主编了表观遗传学领域第一本较为全面的专著Epigenetics,得到了广泛关注。表观遗传学发展实在迅猛,该书于2015年推出第二版。按照该书每8年一版的速度,第三版应该于2023年出版。然而随着Allis的突然离世,不知是否还能等到这第三版Epigenetics。
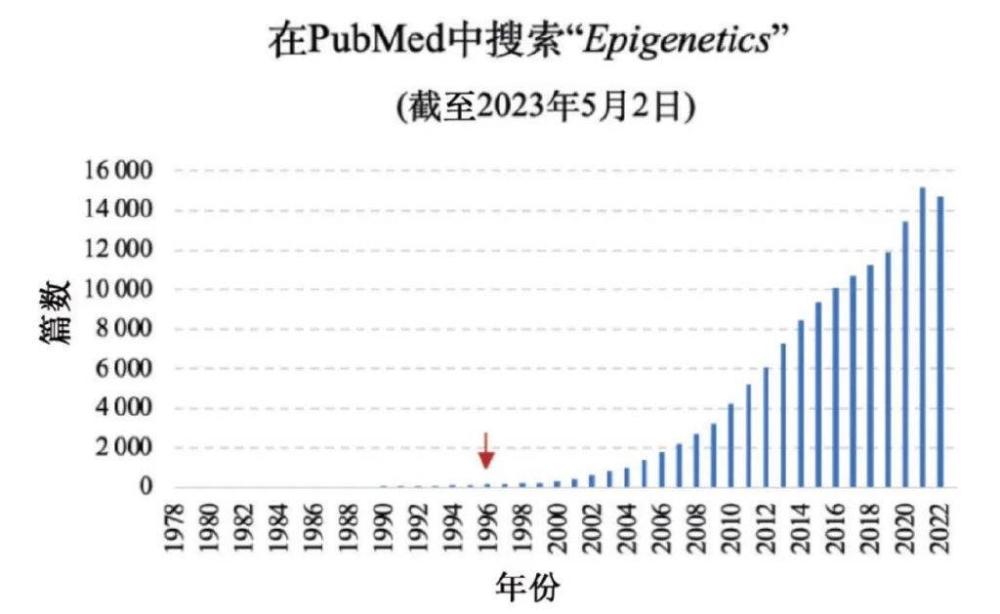
图4 PubMed数据库中的表观遗传相关论文数(截至2023.5.2)
5
Allis独特的科研品味和人格魅力
Allis的科研品味备受赞扬。他长期深耕四膜虫这种独特的模式生物,并借助四膜虫的优势发现第一个组蛋白乙酰基转移酶,便是个绝佳的示例。四膜虫的威力不断显现 (图5)。比如:1963年Ian Read Gibbons在四膜虫中发现第一个动力蛋白 (dynein) [34];1978年Elizabeth Blackburn在四膜虫中发现了端粒[35],并且端粒相关的突破性研究获得2009年诺贝尔生理学或医学奖;1982年Thomas Cech在四膜虫中发现核酸酶 (这一发现更新了人们此前一直认为酶是蛋白质的认知) ,Thomas Cech和Sidney Altman因发现核酶[36-37]而分享了1989年的诺贝尔化学奖。我们无法知道Allis自1978年博后阶段开始研究四膜虫,是否受到1978年在四膜虫中发现端粒的鼓舞。其实在那个年代四膜虫并不热门,时至今日它也远不及小鼠、线虫和果蝇受到关注。Allis勇于解决大问题,不怕冒风险,他相信自己的判断,很多抉择最后被证明是他的远见所在。
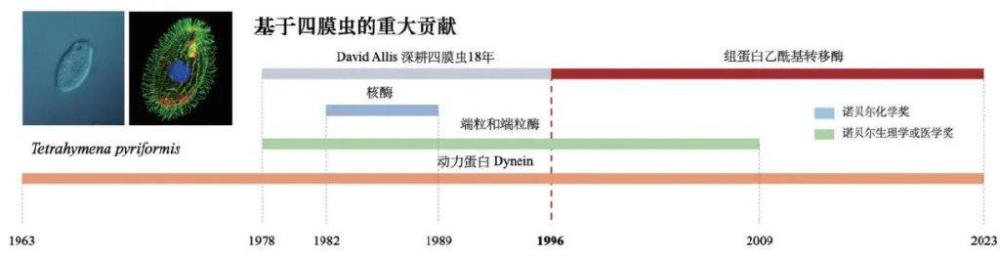
图5 基于四膜虫的重大科研贡献 (颜色条长度可反映时间长度)
很多文章都称赞Allis的科研品味,却忽略了他的坚韧。获得基金支持是开展科研和实验室正常运转的首要前提。Allis提到,在20世纪80年代研究染色质很难获得科研基金,不仅因为缺少研究方法,更重要的在于人们对组蛋白的重要性的认知不足。当时人们认为组蛋白只是结构性蛋白,对于基因转录等重要的生物学过程没有太多的作用。然而Allis坚韧不屈的探索最终掀起了表观遗传学研究的热潮。
Allis非常重视与他人的合作和分享。在组蛋白乙酰基转移酶发现以后,Allis认为,通过分享数据、试剂和想法,科研群体可以一起取得更大的突破。他与Jenuwein的合作便是一个典型的例子。Allis与Jenuwein合作发现了第一个组蛋白甲基转移酶,并一起深入讨论“组蛋白密码”,还一起主编Epigenetics专著。另一个例子是,他制备了H3S10ph的抗体,而且非常慷慨地分享给其他科研人员。至于他从1981年建立自己的实验室以来,先后换了5家研究单位,Shelley Berger (宾夕法尼亚大学表观遗传研究所的创始所长,也是Science悼文的) 认为这与他到处寻求新的合作以及期待与杰出的同事和学生共事有关[4]。
Allis的人格魅力还在于他善于创造有趣和相互帮助的实验室氛围。Mahowald和Gorov sky两个实验室的氛围都非常有趣且友善,让Allis很享受,在他们实验室的工作经历潜移默化地影响了Allis自己实验室的哲学。在Allis建立了自己的实验室以后,他以两位合作导师为榜样,一方面在科研上不断取得突破,另一方面也营造了有趣的实验室氛围。Allis的实验室成员们也表示,即使他们离开了实验室,Allis也会一如既往地支持他们。
Allis的离世让我们深感悲伤,表观遗传学研究领域失去了一位杰出的先驱。然而,他的科学遗产将永远铭刻在表观遗传学的历史中,并将鼓舞着正在路上的研究者们继续前行!
参考文献
[1] DENT S, GREWAL S. C. David Allis (1951–2023) [J]. Nature, 2023, 614(7948): 409.
[2] BERGER S L. C. David Allis (1951–2023) [J]. Science, 2023, 379(6633): 645.
[3] BERNSTEIN E, DENT S, DOU Y, et al. C. David Allis (1951–2023) [J]. Cell, 2023, 186(4): 663-667.
[4] CHI P, LEWIS P W, LU C, et al. Charles David Allis (1951–2023) [J]. Nat Genet, 2023, 55(4): 522-523.
[5] DOWNEY P. Profile of C. David Allis [J]. Proc Natl Acad Sci USA, 2006, 103(17): 6425-6427.
[6] ALLIS C D, GLASER V. Interview with C. David Allis, PhD [J]. Assay Drug Dev Technol, 2015, 13(4): 197-199.
[7] ALLFREY V G, FAULKNER R, MIRSKY A E. Acetylation and methylation of histones and their possible role in the regulation of RNA synthesis [J]. Proc Natl Acad Sci USA, 1964, 51(5): 786-794.
[8] BROWNELL J E, ALLIS C D. An activity gel assay detects a single, catalytically active histone acetyltransferase subunit in Tetrahymena macronuclei [J]. Proc Natl Acad Sci USA, 1995, 92(14): 6364-6368.
[9] BROWNELL J E, ZHOU J, RANALLI T, e t a l . Te t r a h y m e n a h i s t o n e acetyltransferase A: a homolog to yeast Gcn5p linking histone acetylation to gene activation [J]. Cell, 1996, 84(6): 843-851.
[10] MIZZEN C A, YANG X J, KOKUBO T, et al. The TAFII250 subunit of TFIID has histone acetyltransferase activity [J]. Cell, 1996, 87(7): 1261-1270.
[11] O G RY Z K O V V, S C H I LT Z R L , RUSSANOVA V, et al. The transcriptional coactivators p300 and CBP are histone acetyltransferases [J]. Cell, 1996, 87(5): 953-959.
[12] YANG X J, OGRYZKO V V, NISHIKAWA J I, et al. A p300/CBP-associated factor that competes with the adenoviral oncoprotein E1A [J]. Nature, 1996, 382(6589): 319-324.
[13] SPENCER T E, JENSTER G, BURCIN M M, et al. Steroid receptor coactivator-1 is a histone acetyltransferase [J]. Nature, 1997, 389(6647): 194-198.
[14] LIU L, SCOLNICK D M, TRIEVEL R C, et al. p53 sites acetylated in vitro by PCAF and p300 are acetylated in vivo in response to DNA damage [J]. Mol Cell Biol, 1999, 19(2): 1202-1209.
[15] TAUNTON J, HASSIG C A, SCHREIBER S L. A mammalian histone deacetylase related to the yeast transcriptional regulator Rpd3p [J]. Science, 1996, 272(5260): 408-411.
[16] SWEET M T, ALLIS C D. Phosphorylation of linker histones by cAMP-dependent protein kinase in mitotic micronuclei of Tetrahymena [J]. Chromosoma, 1993, 102(9): 637-647.
[17] WEI Y, MIZZEN C A, COOK R G, et al. Phosphorylation of histone H3 at serine 10 is correlated with chromosome condensation during mitosis and meiosis in Tetrahymena [J]. Proc Natl Acad Sci USA, 1998, 95(13): 7480-7484.
[18] WEI Y, YU L, BOWEN J, et al. Phosphorylation of histone H3 is required for proper chromosome condensation and segregation [J]. Cell, 1999, 97(1): 99-109.
[19] REA S, EISENHABER F, O'CARROLL D, et al. Regulation of chromatin structure by site-specific histone H3 methyltransferases [J]. Nature, 2000, 406(6796): 593-599.
[20] KASSIS J A, KENNISON J A, TAMKUN J W. Polycomb and trithorax group genes in Drosophila [J]. Genetics, 2017, 206(4): 1699-1725.
[21] SHI Y, LAN F, MATSON C, et al. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1 [J]. Cell, 2004, 119(7): 941-953.
[22] SCHWARTZENTRUBER J, KORSHUNOV A, LIU X Y, et al. Driver mutations in histone H3.3 and chromatin remodelling genes in paediatric glioblastoma [J]. Nature, 2012, 482(7384): 226-231.
[23] WU G, BRONISCER A, MCEACHRON T A, et al. Somatic histone H3 alterations in pediatric diffuse intrinsic pontine gliomas and non-brainstem glioblastomas [J]. Nat Genet, 2012, 44(3): 251-253.
[24] KHUONG-QUANG D A, BUCZKOWICZ P, RAKOPOULOS P, et al. K27M mutation in histone H3.3 defines clinically and biologically distinct subgroups of pediatric diffuse intrinsic pontine gliomas [J]. Acta Neuropathol, 2012, 124(3): 439-447.
[25] GOLDBERG A D, BANASZYNSKI L A, NOH K M, et al. Distinct factors control histone variant H3.3 localization at specific genomic regions [J]. Cell, 2010, 140(5): 678-691.
[26] LEWIS P W, ELSAESSER S J, NOH K M, et al. Daxx is an H3.3-specific histone chaperone and cooperates with ATRX in replication-independent chromatin assembly at telomeres [J]. Proc Natl Acad Sci USA, 2010, 107(32): 14075-14080.
[27] LU C, JAIN S U, HOELPER D, et al. Histone H3K36 mutations promote sarcomagenesis through altered histone methylation landscape [J]. Science, 2016, 352(6287): 844-849.
[28] NACEV B A, FENG L, BAGERT J D, et al. The expanding landscape of ‘oncohistone’ mutations in human cancers [J]. Nature, 2019, 567(7749): 473-478.
[29] STRAHL B D, ALLIS C D. The language of covalent histone modifications [J]. Nature, 2000, 403(6765): 41-45.
[30] JENUWEIN T, ALLIS C D. Translating the histone code [J]. Science, 2001, 293(5532): 1074-1080.
[31] RANDO O J. Combinatorial complexity in chromatin structure and function: revisiting the histone code [J]. Curr Opin Genet Dev, 2012, 22(2): 148-155.
[32] ALLIS C D, BERGER S L, COTE J, et al. New nomenclature for chromatinmodifying enzymes [J]. Cell, 2007, 131(4): 633-636.
[33] WANG Y, FISCHLE W, CHEUNG W, et al. Beyond the double helix: writing and reading the histone code [J]. Novartis Found Symp, 2004, 259: 3-17.
[34] GIBBONS I R. Studies on the protein components of cilia from Tetrahymena pyriformis [J]. Proc Natl Acad Sci USA, 1963, 50(5): 1002-1010.
[35] BLACKBURN E H, GALL J G. A tandemly repeated sequence at the termini of the extrachromosomal ribosomal RNA genes in Tetrahymena [J]. J Mol Biol, 1978, 120(1): 33-53.
[36] KRUGER K, GRABOWSKI P J, ZAUG A J, et al. Self-splicing RNA: Autoexcision and autocyclization of the ribosomal RNA intervening sequence of Tetrahymena [J]. Cell, 1982, 31(1): 147-157.
[37] GUERRIER-TAKADA C, GARDINER K, MARSH T, et al. The RNA moiety of ribonuclease P is the catalytic subunit of the enzyme [J]. Cell, 1983, 35(3): 849-857.
本文经授权转载自微信公众号“自然撷英”,原题目为《回顾表观遗传学和染色质生物学开拓者C. David Allis传奇的一生》。